Les enzymes sont impliquées dans des processus biologiques essentiels mais leur fragilité est un obstacle à l’obtention de leurs structures intactes par des méthodes conventionnelles de biologie structurale.
Hugo Lebrette (LMGM) et ses collaborateurs ont pu observer pour la première fois la structure atomique de l’état radicalaire, un état extrêmement réactif, d’une enzyme, la ribonucléotide réductase de mycoplasme, grâce à la cristallographie en série couplée à un laser ultra rapide.
Hugo Lebrette et ses collaborateurs ont relevé ce défi grâce au concept de « diffraction avant destruction », en illuminant un flot de cristaux de protéine avec un laser ultra rapide où chaque impulsion dure quelques femtosecondes.
Ce résultat, qui permet une compréhension inédite de la structure d’une enzyme, ouvre la voie à de potentielles innovations thérapeutiques.
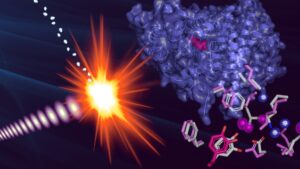 Figure : Structure de la sous-unité R2 de la ribonucléotide réductase de mycoplasme obtenue par cristallographie femtoseconde en série par laser à rayons X à électrons libres. La superposition des acides aminés entre les deux états, radicalaire (rose) et non-radicalaire (gris), montre les changements conformationnels induits par l’acquisition du radical.
Figure : Structure de la sous-unité R2 de la ribonucléotide réductase de mycoplasme obtenue par cristallographie femtoseconde en série par laser à rayons X à électrons libres. La superposition des acides aminés entre les deux états, radicalaire (rose) et non-radicalaire (gris), montre les changements conformationnels induits par l’acquisition du radical.
© Martin Högbom et Hugo Lebrette
Pour en savoir plus :
Communiqué de presse de CNRS-Biologie : https://www.insb.cnrs.fr/fr/cnrsinfo/detection-avant-destruction-un-laser-ultra-rapide-pour-capturer-un-radical-insaisissable
Structure of a ribonucleotide reductase R2 protein radical. Hugo Lebrette, Vivek Srinivas, Juliane John, Oskar Aurelius, Rohit Kumar, Daniel Lundin, Aaron S Brewster, Asmit Bhowmick, Abhishek Sirohiwal, In-Sik Kim, Sheraz Gul, Cindy Pham, Kyle D Sutherlin, Philipp Simon, Agata Butryn, Pierre Aller, Allen M Orville, Franklin D Fuller, Roberto Alonso-Mori, Alexander Batyuk, Nicholas K Sauter, Vittal K Yachandra, Junko Yano, Ville R I Kaila, Britt-Marie Sjöberg, Jan Kern, Katarina Roos, Martin Högbom. Science. 2023 Oct 6;382(6666):109-113. doi: 10.1126/science.adh8160. PMID: 37797025
Contact : Hugo Lebrette


 e quoi s’agit-il ?
e quoi s’agit-il ?







 Le 6 Avril, le LMGM-CBI a reçu des collégiens qui ont participé à la finale régionale du concours «
Le 6 Avril, le LMGM-CBI a reçu des collégiens qui ont participé à la finale régionale du concours «